Die Zukunft der digitalen Pathologie
Revolution der Gewebeanalyse durch Digitalpathologie? Die diagnostische Pathologie erlebt mit der Digitalpathologie momentan einen ähnlich fundamentalen Entwicklungsschub, wie es die Einführung der Histologie Mitte des 19. Jahrhunderts und der Einsatz molekularer Untersuchungen Ende des 20. Jahrhunderts waren.
Whole Slide Imaging: die Basis für Telepathologie und maschinelle Bildanalyse
Die Begriff „Digitalpathologie“ umfasst eine breite Palette von Bildgebungstechniken und Informationsverarbeitungsmethoden in der Pathologie. Entscheidender Meilenstein in der Geschichte der Digitalpathologie war die Entwicklung digitaler Mikroskope, durch welche erstmals histologische Schnitte in ihrer Gesamtheit in hoher Auflösung gescannt und digitalisiert werden konnten, sogenannte „whole slide images“ (WSI). Deren kommerzielle Verfügbarkeit läutete vor ca. 20 Jahren eine neue Ära in der Pathologie ein. Hierdurch wurden histologische Schnitte in Echtzeit und mikroskopunabhängig auch über große Distanzen hinweg verfügbar gemacht. Diese als „Telepathologie“ bezeichnete Technologie vereinfachte Konsultationen zwischen Patholog:innen und ermöglichte einen rascheren Zugang zu spezifischer pathologisch-diagnostischer Expertise. Auch in der Lehre werden WSI häufig eingesetzt, um Studierenden eine interaktive und visuell ansprechendere Darstellung histologischer Präparate beim Selbststudium zu Hause zu ermöglichen.
In den ersten Jahren waren die diagnostische und wissenschaftliche Telepathologie sowie die medizinische Lehre die dominanten Einsatzgebiete der WSI. Zugleich wurden sie auch die Basis für die Entwicklung von Methoden zur maschinellen Bild- und Gewebeanalyse.
Von der Histomorphometrie zum Deep Learning: digitale Bildanalyse in der Pathologie
In den ersten Jahren befasste sich die maschinelle Bildanalyse in der Digitalpathologie vorrangig mit der Segmentierung von histologischen Strukturen. Hierbei handelt es sich um eine automatische oder halbautomatische Identifizierung und Abgrenzung von verschiedenen Gewebearten und Organstrukturen bis hin zu einzelnen Zellen in digitalen Histologiebildern. Diese Segmentierung bildet die Grundlage für quantitative und qualitative Analysen von Gewebeproben (sog. Histomorphometrie).
Bei diesen Analysen kamen initial Techniken der sogenannten klassischen Bildanalyse (d. h. Computer Vision) zum Einsatz. Diese waren jedoch aufgrund der Komplexität histomorphologischer Strukturen oftmals auf die Erkennung und Beschreibung von Zellkernen beschränkt. Jedoch kamen diese Methoden bereits vor mehr als 25 Jahren zur Erforschung z. B. des Tumor-Gradings zum Einsatz.1 Weiters fanden sie Anwendung in der maschinellen Auswertung von Immunhistochemie-(IHC-) Bildern, wofür inzwischen eine Fülle von für die diagnostische Anwendung zertifizierten Tools kommerziell verfügbar ist (z. B. für die Expression von HER2, des Proliferationsmarkers Ki-67 und von Östrogen- bzw. Progesteronrezeptoren beim Mammakarzinom).
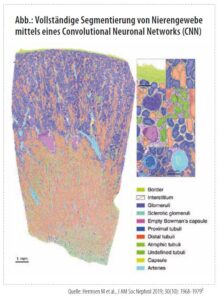
Jedoch erst die rapide Entwicklung des maschinellen Lernens sowie der Mustererkennung durch künstlich intelligente Systeme (KIs) führte zu einem exponentiellen Fortschritt in der Digitalpathologie innerhalb des letzten Jahrzehnts. Diese neuen Methoden konnten zum Beispiel bereits für die Segmentierung immer komplexerer histologischer Muster (wie in der Arbeit von Meyke Hermsen et al. in der Abbildung dargestellt)2, die Detektion von Lymphknoten-Metastasen3 oder das Gleason-Grading von Prostata-Biopsien4 eingesetzt werden.
Darüber hinaus zeigt die rezenteste Forschung, dass solche Systeme auch eingesetzt werden, um Aufgaben zu lösen, die Patholog:innen unzugänglich sind, z. B. die Vorhersage des Mikro-Satelliten-Status in kolorektalen Karzinomen rein aus der Histomorphologie, ohne genetische Tests.5
Diese Beispiele zeigen das hohe Potenzial solcher Systeme im Bereich der Digitalpathologie. Jedoch stellen sie auch neue Herausforderungen dar. So benötigen moderne KIs für das adäquate Lernen eine hohe Fallzahl an repräsentativen und kuratierten Bildern (oftmals mehrere tausend oder zehntausend). Weiters sind diese Systeme derzeit noch hochspezialisiert und agnostisch gegenüber pathologischem Vorwissen und dadurch in ihrem Einsatz auf Patholog:innen angewiesen.
Digital Spatial Profiling (DSP)
Digital Spatial Profiling (DSP) ist eine neuartige Technologie in der Digitalpathologie, die Bildgebungsverfahren mit molekularer Analytik kombiniert, um die Lokalisation und Konzentration verschiedener Moleküle (wie DNA, RNA und Proteine) direkt in Gewebeschnitten zu erfassen. DSP basiert auf der Verwendung von fluoreszenzmarkierten Sonden, die spezifisch an Zielmoleküle wie Proteine oder RNA-Moleküle binden. Ein Lasersystem scannt die Probe und misst die Stärke der Fluoreszenzsignale, die Aufschluss über Menge und genaue Lokalisation der Zielmoleküle geben. Die große Stärke von DSP liegt in der Kombination von räumlicher Information mit hochgradiger Multiplex-Fähigkeit, d. h. es können viele verschiedene Moleküle simultan in situ analysiert werden. Dies ermöglicht es, komplexe biologische und pathologische Zusammenhänge besser zu verstehen.
Mikroskopische Gewebeuntersuchung ohne histologische Gewebeaufarbeitung
Die oben genannten Verfahren beruhen auf einer Digitalisierung von konventionellen histologischen Schnitten. Anders als in der Radiologie, wo primär digitale Bilder generiert werden, folgt die Digitalisierung in der Histologie der konventionellen Gewebeaufarbeitung und ist damit ein zeitlich und technisch sehr aufwändiger Prozess.
Ex Vivo Confocal Microscopy (ECM) und Raman Microscopy (RM) sind fortschrittliche Bildgebungstechnologien, die eine mikroskopische Gewebeanalyse ohne vorherige histologische Aufarbeitung ermöglichen.
- Bei der ECM werden unfixierte Gewebeproben mit Fluoreszenzfarbstoffen zur Markierung von Zellkernen inkubiert, und mittels Laser-Scanning-Mikroskop werden digitale Bilder in hoher Auflösung und Tiefenschärfe generiert. Digital kann eine Kolorierung erzeugt werden, die der vertrauten Hämatoxilin-Eosin-Färbung konventioneller Histologieschnitte sehr nahekommt.
- Raman-Mikroskopie ist eine spektroskopische Technik, die auf dem Raman-Effekt basiert. Sie beruht auf der Streuung von Licht, das auf ein Molekül trifft und dabei seine Energie ändert, je nach den spezifischen Schwingungen und Rotationen der Moleküle. Diese Änderungen im Lichtspektrum liefern Informationen über die Zusammensetzung und Struktur der untersuchten Materialien und werden verwendet, um digitale Schnittbilder aus unbehandelten Gewebeproben zu generieren, die analog zu den Bildern der ECM digital eingefärbt werden.
Beide Verfahren erlauben eine rasche, zerstörungslose Generierung primär digitaler mikroskopischer Bilder im Sinne virtueller Gewebeschnitte. Dies prädestiniert sie für den Einsatz in der intraoperativen mikroskopischen Diagnostik.
Deep-Learning-Modell DeepGlioma: Einen faszinierenden Ausblick auf das außerordentliche Potenzial der Kombination von digitaler Mikroskopie mit Deep-Learning-Methoden gewährt eine rezente Studie über die diagnostische Klassifikation von Hirntumoren.6 Die Autoren stellen ein spezielles Deep-Learning-Modell (DeepGlioma) vor, das mittels eines multimodalen Datensatzes trainiert wurde, der auf RM und umfangreichen öffentlichen Genomdaten basiert. In einer prospektiven, multizentrischen Testkohorte von Patient:innen mit diffusem Gliom konnte DeepGlioma die molekularen Veränderungen vorhersagen, die von der WHO zur Definition des diffusen Glioms verwendet werden.
Diese Strategie hat mehrere wichtige Aspekte: Sie kommt ohne die konventionelle, sehr umfangreiche multimodale Diagnostik (Histologie, IHC, Sequenzierung) aus und verkürzt die Dauer des Verfahrens so sehr, dass es auch intraoperativ eingesetzt werden könnte.
Aktuelle und zukünftige diagnostische Anwendungen der Digitalpathologie
Die Digitalpathologie im Allgemeinen beziehungsweise Methoden der maschinellen Gewebeanalyse im Speziellen haben großes Potenzial, sowohl die Arbeit von Patholog:innen zu erleichtern als auch die Präzision der Diagnostik zu erhöhen. Maschinelle Gewebeanalyse kann sicher in manchen Bereichen nicht nur schnellere, sondern auch präzisere Resultate generieren und potenziell auch zur Identifikation neuer diagnostischer Indikatoren in digitalen Gewebeschnitten führen.7
Obwohl die technische Basis der digitalen Bilderstellung in der Pathologie bestens etabliert und eine Vielzahl von erprobten Systemen kommerziell erhältlich ist, wird die Digitalpathologie nur zögerlich beziehungsweise im geringen Umfang im diagnostischen Alltag eingesetzt. Dies hat mehrere Gründe: Ein wesentliches Hindernis ist die mangelnde routinemäßige Verfügbarkeit von WSI. Dies scheitert weniger am Zugang zu geeigneten Scannern, sondern vielmehr am Fehlen eines integrierten, digital pathologischen Workflows. Dieser würde eine enge Integration von histologischer Gewebeaufarbeitung, Laborinformationssystem und Digitalisierungsservice bedingen, deren Herstellung jedoch sowohl technisch als auch organisatorisch aufwändig und mit hohen Kosten verbunden ist. Auch haben innovative technische Plattformen wie DSP oder ECM beziehungsweise RM zum Teil sehr hohe Anschaffungs- oder Betriebskosten.
Die mangelnde systematische Verfügbarkeit von WSI behindert nicht nur die Integration bestehender digitaler Technologien in den diagnostischen Alltag, sondern erschwert auch die Entwicklung neuer Modelle zur Gewebeanalyse, da diese häufig auf der Analyse sehr großer Mengen von (meist auch gut annotierten) digitalen Bilddaten beruhen.
Nicht zuletzt ist auch eine gewisse Zögerlichkeit von Patholog:innen zu beobachten, die neuen Technologien anzunehmen, die auch eine deutliche Änderung des Arbeitsablaufes mit sich bringen.
Da die Vorteile der neuen Technologien für die Geschwindigkeit und die Qualität der histologischen Diagnostik evident sind und die Verfahren auch deutliche Erleichterungen für den Arbeitsablauf von Patholog:innen mit sich bringen, muss dringend gewährleistet werden, dass ein digitaler Workflow zur Erstellung von WSI in möglichst vielen Abteilungen etabliert wird, um so die Basis für die Implementierung vorhandener Anwendungen und für die Entwicklung neuer maschineller Analysemethoden zu schaffen.
Referenzen: (1) Choi HK et al., Anal Cell Pathol 1997; 15(1):1–18 (2) Hermsen M et al., J Am Soc Nephrol 2019; 30(10):1968–79 (3) Bandi P et al., IEEE Trans Med Imaging 2019; 38(2):550–60 (4) Bulten W et al., Nat Med 2022; 28(1):154–63 (5) Wagner SJ et al., Cancer Cell 2023; 41(9):1650–61.e4 (6) Hollon T et al., Nat Med 2023; 29(4):828–32 (7) Verghese G et al., J Pathol 2023; 260(5):551–63