Biomarker aus dem Umfeld chronischer Nierenerkrankungen
Die nephrologische Labordiagnostik bietet eine Palette von gut dokumentierten Parametern für Erst- und Differenzialdiagnostik, und mit Einschränkungen auch für die Prognose (Hofmann W, J Lab Med 2011; 35:127–146).
Die verwendeten Marker sind großteils schon sehr lange in Verwendung – die Kreatininbestimmung wurde 1917 beschrieben, und neue Marker finden nur sehr langsam den Weg in die Routine. Dieser Artikel soll diese Situation zu erklären versuchen und einen Ausblick auf laufende Entwicklungen und deren Potenzial geben.
Grundlegende Fortschritte in Genomik und Proteomik eröffnen neue Perspektiven. Translationale Forschung, also die Kombination von Zellbiologie und Physiologie, Epidemiologie, klinischen Beobachtungen und Studien – verspricht, unser diagnostisches Repertoire signifikant zu verbessern und zu erweitern. Zwar sind seit Beginn des Human Genome Project mehr als 20 Jahre vergangen, und seit bald 10 Jahren ist das humane Genom komplett sequenziert. Der ersten Euphorie über diese Errungenschaft folgte eine Phase der Konsolidierung, und erst allmählich ergeben sich aus diesen Erkenntnissen auch praktische Konsequenzen (Edwards AE, Nature 2011; 470:163–5).
Die klinische Entität als Maßstab für einen neuen Marker
Die Erwartungen an einen optimalen Biomarker sind offensichtlich und begründen sich auf Physiologie, Analytik/Praktikabilität und Klinik: biologisch plausibel, nichtinvasiv, präzise und einfach zu messen, weiter dynamischer Bereich, sensitiv, spezifisch, prädiktiv und theranostisch. Trotz dieser sehr klaren Kriterien ist es ein langwieriger Prozess, ein etabliertes Schema der IVD zu adaptieren. Einerseits gibt es das verständliche und berechtigte Beharren auf etablierten Prozessen, besonders in einer Situation finanzieller und organisatorischer Verknappung. Andererseits stehen wir auch vor einem statistischen Problem. Eine korrekte Beurteilung des Informationsgehaltes eines Laborwertes – das Gleiche gilt natürlich für jede Art von Information oder Symptom – ist nur in Kenntnis der tatsächlichen Diagnose und damit oft erst retrospektiv möglich. Alternativ wird gern ein Vergleich mit etablierten Markern herangezogen, mit denen es langjährige Erfahrung gibt, was aber auch irreführend sein kann. Dazu eine ernüchternde quantitative Analyse (Waikar SS et al, JASN 2012; 23:13–21): Bei 80 % Sensitivität und 90 % Spezifität (typische Leistungsmerkmale für die Kreatinin-Bestimmung) und einer Prävalenz von 10 % würde ein „perfekter“ Nierenmarker (100 % Sensitivität und 100 % Spezifität) im Vergleich zu Kreatinin nur eine Spezifität von 47 % ergeben – also fälschlicherweise deutlich schlechter beurteilt werden.
 Die korrekte intrinsische Qualität eines neuen Markers kann daher nur in großen Studien mit Verlaufsproben angemessen erhoben werden, in denen eben der „golden standard“ nicht ein unvollkommener Laborwert, sondern die reale klinische Entität der Maßstab ist. In der Praxis bedeutet das ein grundlegendes Umdenken in der Evaluierung und zugleich eine erhebliche Hürde. Eine evidenzbasierte Leistungsbewertung soll weitgehend auf klinischen Daten basieren und nicht nur in Korrelation zu den bekannten Laborparametern.
Die korrekte intrinsische Qualität eines neuen Markers kann daher nur in großen Studien mit Verlaufsproben angemessen erhoben werden, in denen eben der „golden standard“ nicht ein unvollkommener Laborwert, sondern die reale klinische Entität der Maßstab ist. In der Praxis bedeutet das ein grundlegendes Umdenken in der Evaluierung und zugleich eine erhebliche Hürde. Eine evidenzbasierte Leistungsbewertung soll weitgehend auf klinischen Daten basieren und nicht nur in Korrelation zu den bekannten Laborparametern.
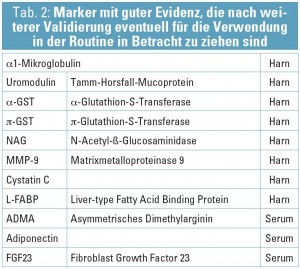
Im Laufe der letzten Jahre wurden einige spezifische Marker solchen rigorosen Studien unterzogen und sind daher ernsthafte Kandidaten für routinemäßige Anwendung (siehe Tab. 1; Sirota JC, et al., J Toxicol 2011; 328120; Devarajan P, Adv Chronic Kiney Dis 2010; 17: 469–479). Diese Liste wird laufend durch die verbesserte und verfeinerte Analytik des humanen Proteoms erweitert (Konvalinka A, Clin Chem 2012; 58: 353–365, Mischak H et al., Proteomics Clin Appl 2011; 5: 211). Wesentliche Kriterien sind natürlich, dass der Analyt routinemäßig behandelt werden kann, also keine spezielle Abnahme erfordert, beim Transport stabil ist, keinen ausgeprägten zirkadianen Rhythmus zeigt und bei Harn als Probenmatrix das Ergebnis einfach und korrekt auf Kreatinin normiert werden kann.
Die Problematik anhand von FgF23 illustriert
Einer der neueren Marker (aus Tabelle 2), dessen (patho) physiologische Funktion ausreichen gut untersucht ist, um daraus erhebliches diagnostisches und auch therapeutisches Potenzial abzuleiten, ist das in den Osteozyten produzierte FGF23 (In Nephroskript 1/2011: S14–17 findet sich dazu eine rezente Übersicht von Ribisch und Rosenkranz).
Hier soll anhand der Bestimmung von FGF23 gezeigt werden, welche Fragen hinreichend zu beantworten sind, bevor eine routinemäßige Verwendung empfohlen werden kann (Wolf M, JASN 2010; 1427–35; Isakova T et al., JAMA 2011; 305:2432–9; Wessley-Perry K, Clin Chem 2011; 11:1476–7).
- Zirkulierendes FGF23 ist heterogen, derzeit verfügbare Immunoassays erkennen das intakte Protein bzw. ein Cterminales Fragment. Welche Fragmente sind diagnostisch relevant und zugleich praktikabel? Erste publizierte Studien legen nahe, dass die Ergebnisse dieser unterschiedlichen Methoden gut korrelieren.
- Es fehlen gut dokumentierte Referenzbereiche für Normalpersonen, Alters- und Geschlechtsabhängigkeit (Phosphatspiegel sind deutlich altersabhängig, Östradiol stimuliert FGF23).
- Welchen Einfluss hat die Phosphataufnahme?
- Welche Erkrankungen außer einer eingeschränkten Nierenfunktion sind mit erhöhten FGF23-Spiegeln assoziiert?
- Welche Korrelation besteht im Detail zwischen FGF23 und dem Grad der Niereninsuffizienz?
- In welchem Zusammenspiel stehen FGF23 und PTHSpiegel? Es gibt zahlreiche Studien zu PTH, der Bedeutung bestimmter Fragmente (d. h. der Spezifität der Messmethoden) und den entsprechenden Referenzbereichen. Für FGF23 fehlen solche Daten fast vollständig.
- Wie wird FGF23 reguliert? Sind andere Faktoren (Klotho, DMP-1, MEPE, PHEX) direkter mit der Hyperphosphatämie assoziiert; oder gibt es einen noch nicht identifizierten renalen Faktor, der eine beginnende Phosphatbelastung erkennt und die Sekretion von FGF23 steuert?
- Hat FGF23 in den extrem hohen pathologischen Konzentrationen auch unerwartete und unerwünschte Wirkungen auf andere Gewebe?
- Ist FG23 theragnostisch?
 Dieses Beispiel illustrieren, welchen Fragen sich alle weiteren „neueren“ Marker stellen müssen, ehe sie auch nur als Kandidaten für die Routine in Betracht gezogen werden können. Auch wenn IVD-Marker nur Surrogate darstellen, ähnelt eine hinreichende Leistungsbewertung konzeptuell pharmakologischen – und damit natürlich wesentlich rigoroseren – Studien. Im Verlauf einer solchen Bewertung wird auch klar werden, welche zusätzlichen Informationen sich aus der Bestimmung eines solchen Markers ergeben, und ob sein Einsatz nicht sogar gefordert werden muss. Ein illustratives Beispiel ist die verbesserte Diagnostik durch die Einführung von CK-MB, Troponin und (NT-pro) BNP in konsekutiven Wellen in der Kardiologie. Anzumerken ist auch noch, dass der Informationsgehalt dieser Bestimmungen immer weiter verbessert wird, einerseits durch genauere Auswertungen einer immer weiter wachsenden Anzahl von Studien, aber auch durch spezifischere und empfindlichere Messmethoden.
Dieses Beispiel illustrieren, welchen Fragen sich alle weiteren „neueren“ Marker stellen müssen, ehe sie auch nur als Kandidaten für die Routine in Betracht gezogen werden können. Auch wenn IVD-Marker nur Surrogate darstellen, ähnelt eine hinreichende Leistungsbewertung konzeptuell pharmakologischen – und damit natürlich wesentlich rigoroseren – Studien. Im Verlauf einer solchen Bewertung wird auch klar werden, welche zusätzlichen Informationen sich aus der Bestimmung eines solchen Markers ergeben, und ob sein Einsatz nicht sogar gefordert werden muss. Ein illustratives Beispiel ist die verbesserte Diagnostik durch die Einführung von CK-MB, Troponin und (NT-pro) BNP in konsekutiven Wellen in der Kardiologie. Anzumerken ist auch noch, dass der Informationsgehalt dieser Bestimmungen immer weiter verbessert wird, einerseits durch genauere Auswertungen einer immer weiter wachsenden Anzahl von Studien, aber auch durch spezifischere und empfindlichere Messmethoden.
Hoffnungsträger aus der Genomforschung
Eine weitere Gruppe von „explorativen“ Markern ergibt sich aus unserer Kenntnis des gesamten humanen Genoms mit seinen ca. 22.000 Genen – etwas weniger als erwartet – und ca. 300.000 davon abgeleiteten Genprodukten. Aus Genexpressiondaten auf mRNA-Ebene wissen wir über die gewebespezifische Expression bestimmter Gene, wodurch eine Gruppe von interessanten Targets identifiziert werden konnte. Darüber hinaus bietet sich ein guter, wenn auch nicht vollständiger Überblick über die vernetzten Stoffwechselwege – aus dem sich gewebespezifische Knotenpunkte ableiten lassen, die ebenfalls Kandidaten für Biomarker sind. Eine solche In-silico-Auswahl kann durch publizierte Studien, Zellkultur- und Tierexperimente ergänzt und anhand von Stoffwechselwegen annotiert werden. Eine zusätzliche Quelle sind GWAS (genome wide association studies), die durch Vergleich der gesamten DNA von Erkrankten und gesunden Kontrollpersonen Hinweise auf involvierte Gene liefern. Die Kombination dieser Methoden liefert Hinweise auf eine Vielzahl von Genen und deren Produkten, die Zusammenhang mit spezifischen Pathologien stehen könnten (Li Y, Shiffman D, Oberbauer R., Methods Mol Biol 2011; 719: 219–34). Aus einer primären Liste von Kandidaten qualifizieren sich dann einzelne, für die sich ergänzende Begründungen für eine wichtige Rolle in der Genese renaler Erkrankungen ableiten lassen. Ein Vorteil dieses gezielten Vorgehens ist es, solche Biomarker zu untersuchen, die spezifisch einzelne Prozesse oder Zelltypen der Nierenfunktion abbilden, wie die Filtrationsleistung der Podozyten, vaskuläre Läsionen, fibrotische Veränderungen oder lokale entzündliche Prozesse. Eine Reihe solcher Marker, deren diagnostisches Potenzial hoch genug scheint, um damit eine Validierung zu beginnen, bzw. – soweit noch nicht verfügbar – zuerst entsprechende Messmethoden zu entwickeln, ist derzeit Forschungsgegenstand im Rahmen des SysKid- Projekts.
Mehr Information durch Kombination von Markern
Das beste Material für Favoriten aus dieser Gruppe sind Probenpanels aus retrospektiven klinischen Studien, die es ermöglichen, den prognostischen Wert und den Einfluss therapeutischer Maßnahmen auf diese Marker zu beurteilen. Kollektive wie jenes der berühmte Framingham-Studie, aber auch lokale Probensammlungen (Bruneck-Studie) sind besonders wertvoll, um die praktische Qualität solcher theoretischer Marker zu beurteilen. Weitere Fortschritte sind von einer verbesserten Präsentation der Ergebnisse zu erhoffen. Einfache Referenzbereiche sind zwar anschaulich, dieser dichotome Zugang verzichtet aber auf einen großen Teil der Information. Algorithmen, durch die Zusammenhänge verschiedener Parameter über den gesamten Bereich gut interpretierbar dargestellt werden, sollten – auch durch die Kombination mehrerer Marker – verbesserte diagnostische Information liefern.
In letzter Konsequenz ist die Praktikabilität eines Biomarker entscheidend, also ob dieser präanalytisch robust und damit routinetauglich ist.