Proteomik – Was werden Proteomanalysen in Zukunft therapeutisch bringen?
Proteomik ist ein junger, moderner Forschungsbereich, der sich mit den wesentlichen Bausteinen des Lebens, den Proteinen, auseinandersetzt. Die wichtigsten Werkzeuge dafür im Labor sind analytische Geräte mit unglaublicher Empfindlichkeit und Analysegeschwindigkeit, so genannte Massenspektrometer. In Kombination mit aufwändiger bioinformatischer Computer-Unterstützung ist es mit diesen Werkzeugen möglich, die Identität und Konzentration von hunderten bis tausenden von verschiedenen Proteinen in einer Probe zu bestimmen1.
Multiples Myelom
Das multiple Myelom ist für die Grundlagenforschung eine Erkrankung mit Modellcharakter und somit von großer Bedeutung. Eine Krebserkrankung kann generell nicht mehr als zufällige Gendefekterkrankung von einzelnen, sich dann hemmungslos vermehrenden Krebszellen begriffen werden. Vielmehr stellt sie eine komplexe Abfolge von versagenden Regulationsprozessen dar, an deren Ende zwar ein Zelltyp zu Tumorzellen transformiert, aber bei der verschiedenste andere Zelltypen schon in frühen Stadien der Erkrankung funktionell beteiligt sind. Beim multiplen Myelom sind B-Lymphozyten transformiert, die ursprünglich im Knochenmark gebildet werden. Von besonderer Bedeutung ist hier die klinische Darstellung einer nichtmalignen Vorerkrankung (Präneoplasie), dem MGUS (monoklonale Gammopathie unklarer Signifikanz), das der eigentlichen Tumorerkrankung wahrscheinlich immer vor – ausgeht2, oft aber unerkannt bleibt. Die B-Lymphozyten im MGUS tragen schon alle wesentlichen genetischen Aberrationen, die für das multiple Myelom charakteristisch sind3. Ein Forscherteam an der Klinik für Innere Medizin I unter der Leitung von Prof. Johannes Drach hat in aktueller Forschungsarbeit in Zusammenarbeit mit dem Institut für Krebsforschung unter Einsatz von Proteomanalysen festgestellt, dass fibroblastenähnliche mesenchymale Vorläuferzellen aus dem Knochenmark ganz charakteristische Veränderungen in ihrem Funktionsprofil beim Übergang vom MGUS zum multiplen Myelom aufweisen (Slany et al., Manuskript in Vorbereitung). Diese Veränderungen zeichnen sich insbesondere dadurch aus, dass diese genetisch nicht veränderten Zellen durch die erhöhte Sekretion von Wachstumsfaktoren und veränderter extrazellulärer Matrix-Proteine (ECM-Proteine) die transformierten Lymphozyten in ihrem Wachstum unterstützen und sie sogar gegen den Angriff von Immunzellen bzw. durch Chemotherapeutika schützen können. Wir konnten somit feststellen, dass genetisch nicht veränderte Stromazellen den Tumorzellen ganz entscheidende Unterstützung zukommen lassen und somit auch ein Ziel einer kombinierten Therapieform darstellen sollten. Für das multiple Myelom könnte es damit möglich werden, durch eine gezielte therapeutische Intervention den körpereigenen tumorpromovierenden Effekt der Stromazellen zu reduzieren und somit die Tumorzellen empfindlicher gegen eine nachfolgende zytostatische Therapie zu machen.
Klinische Proteomik
In weiterer Folge soll ausgeführt werden, wie die Proteomik-Technologie zur verbesserten Diagnostik, Stratifizierung von Patienten für eine optimale Therapiewahl als auch für die Entwicklung neuer Therapieformen beitragen kann. Wie kann eine rein analytische Methodik überhaupt Bedeutung für die Medizin erlangen und zur besseren Diagnostik und sogar Therapie-Effizienz etwa von Myelom- Patienten beitragen? Tatsächlich kann man diese Frage nur in Betrachtung der biologischen Bedeutung von Proteinen schlüssig beantworten.
Proteine sind die wichtigsten funktionellen Bausteine des Lebens. Sie bewerkstelligen fast alle Funktionen, die für das Leben charakteristisch sind, wie etwa Organisation und Aufrechterhaltung der räumlichen Struktur von Zellen und Geweben, Metabolismus, Fähigkeit zur Vermehrung, zur Aufnahme und Verarbeitung von Informationen und vieles mehr. Man kann sich Proteine wie Bauelemente und Werkzeuge vorstellen, wie Mikromaschinen, die ganz spezifische Aufgaben erledigen können. Interessanterweise ist das zur Verfügung stehende Repertoire zwar recht groß, aber trotzdem durch das Genom definiert und somit limitiert. Ein Mensch kann etwas mehr als 20.000 unterschiedliche Proteine herstellen, wobei jeder Mensch im Wesentlichen über genau das gleiche Repertoire verfügt. Dieses ist auch der Werkzeug-Ausstattung etwa von Säugetieren verblüffend ähnlich. Das Geheimnis dürfte darin liegen, dass ein bestimmtes Werkzeug wie etwa ein Meißel in der Hand eines Bildhauers ganz andere Resultate hervorbringen kann als in der Hand eines Bauarbeiters. Der Analytiker kann nun sehr gut feststellen, dass im Verlauf eines biologischen Prozesses bestimmte Zellen ein Werkzeug wie etwa einen „Meißel“ verstärkt einsetzen, wofür dieses aber letztlich eingesetzt wird, kann oft nur aufgrund von viel Verständnis und Erfahrung abgeschätzt werden.
Die Analyse jener Proteine, die im Verlauf einer Erkrankung verstärkt zum Einsatz kommen, kann somit zu einem verbesserten Verständnis dieser Erkrankung beitragen, ist aber nie trivial zu interpretieren. Es ist notwendig, ein sehr genaues Verständnis von Zellen und deren biologischen Funktionen zu haben, bevor man krankheitsbedingte Veränderungen im Kontext der Bildung spezifischer Proteine überhaupt verstehen kann.
Zellen als Produzenten der Proteine
Zellen sind die kleinsten potenziell autonomen Lebenseinheiten, die unseren Körper bilden. Zellen sind auch die hierarchische Ebene, auf der entschieden und ausgeführt wird, welche und wie viele Proteine gebildet werden. Man kann vielleicht den menschlichen Körper in Analogie zu einem Staat verstehen, in dem bestimmte „Staatsorgane“ wie Legislative und Exekutive oder etwa auch die Industrie spezifische Funktionen zu erfüllen haben, so wie im Körper das Gehirn, die Muskeln oder die Leber unterschiedliche Funktionen erfüllen. Innerhalb eines Staatsorganes gibt es spezialisierte Fachkräfte wie Rechtsanwälte, Kaufleute, Mechaniker oder Bioinformatiker. Diese bedienen sich vornehmlich bestimmter Werkzeuge, die charakteris – tisch für ihre Berufsausübung sind, wie Gesetzesbücher, Werbemittel, Maschinen oder Computer. Diese Fachkräfte und Werkzeuge wiederum können in Analogie zu spezialisierten Zelltypen gesehen werden, wie etwa Nervenzellen, Gefäßzellen, Muskelzellen oder Leberzellen, die sich charakteristischer Proteine als ihrer typischen Werkzeuge bedienen. Wenn sie ein Gebäude betreten, können sie allein anhand der darin befindlichen Ausstattung und Werkzeuge in etwa abschätzen, wozu dieses Gebäude benutzt wird, sei es als Wohnhaus, Schule oder Industriewerkstatt. So geht es einem Bioanalytiker bzw. Proteomiker mit einem experimentellen Resultat aus einer massenspektrometrischen Analyse. Er kann erkennen, welche Werkzeuge in einer bestimmten Situation gerade zum Einsatz gekommen sind, aber er kann den eigentlichen Gebrauch der Werkzeuge nicht direkt beobachten, sondern nur erahnen.
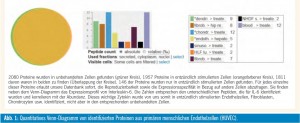
Aus diesem Grund müssen wir viele Referenzen in detaillierter Kleinarbeit herstellen und analysieren. Wir isolieren zum Beispiel einen definierten Zelltyp wie etwa Endothelzellen und bringen ihn durch einen definierten Reiz wie etwa entzündliche Stimulierung dazu, einephysiologisch relevante Tätigkeit aufzunehmen. Vergleichende Proteomanalysen dieser Zellen vor und nach Aufnahme der Tätigkeit können es uns ermöglichen, jene „Werkzeuge“ zu identifizieren, die charakteristisch für die untersuchte Tätigkeit sind (Abb. 1). Wir haben in den letzten Jahren von vielen hunderten derart geplanten Refenzexperimenten alle Daten in einer großen Datenbank gesammelt und können somit für tausende Proteine sofort bestimmen, ob sie für bestimmte Zelltypen oder bestimmte Tätigkeiten charakteristisch sind4.
Entwicklung einer Forschungsstrategie
 Kommen wir wieder zum multiplen Myelom zurück, für das wir ja verbesserte, individualisierte Therapieformen entwikkeln wollen. Eine vergleichende Analyse der mesenchymalen Vorläuferzellen aus dem Knochenmark von gesunden Spendern (Hüftgelenksoperationen) mit den entsprechenden Zellen aus Myelomzellen hat ergeben, dass diese Zellen Faktoren ausschütten, welche die Krankheit fördern können. Daraus ergibt sich zwingend die Frage, welcher Reiz diese Zellen dazu gebracht hat, diese nicht erwünschte Tätigkeit aufzunehmen. Wir untersuchen das, indem wir die Zellen von gesunden Spendern in vitro in unterschiedlicher Weise stimulieren und mittels nachfolgender Proteomanalysen untersuchen, ob die Proteine gebildet wurden, die wir sonst nur in den Zellen aus erkrankten Patienten gefunden haben (Abb. 2). So konnten wir etwa feststellen, dass eine entzündliche Aktivierung zumindest einen Teil der ungewünschten Aktivitäten verursachen kann. Wenn wir einen Eindruck davon erhalten haben, welche Funktionen letztlich für den krankhaften Zustand verantwortlich sind, können wir gezielt auf die Suche nach Wirkstoffen gehen, die diese ungewünschten Funktionen unterdrücken. Hier zeigt sich jetzt ganz besonders die Nützlichkeit von umfassenden Proteomanalysen. Hat man erst einmal die relevanten Fehlfunktionen erkannt und analysiert, kann man Markerproteine bestimmen, welche diesen Zustand indizieren können. Diese Markerproteine können somit für diagnostische Zwecke herangezogen werden. Gleichzeitig können diese Markerproteine auch eingesetzt werden, um in In-vitro-Studien geeignete Wirkstoffe zu finden, welche diese ungewünschten Aktivitäten unterbinden können. Schließlich können dieselben Proteine auch analysiert werden, um im Patienten auch nachzuvollziehen, ob eine eingeleitete Therapie auch wirklich den gewünschten Erfolg erbringt. Nur wenn die Markerproteine im Blut des Patienten zurückgehen, hat das Medikament den gewünschten Erfolg erzielt.
Kommen wir wieder zum multiplen Myelom zurück, für das wir ja verbesserte, individualisierte Therapieformen entwikkeln wollen. Eine vergleichende Analyse der mesenchymalen Vorläuferzellen aus dem Knochenmark von gesunden Spendern (Hüftgelenksoperationen) mit den entsprechenden Zellen aus Myelomzellen hat ergeben, dass diese Zellen Faktoren ausschütten, welche die Krankheit fördern können. Daraus ergibt sich zwingend die Frage, welcher Reiz diese Zellen dazu gebracht hat, diese nicht erwünschte Tätigkeit aufzunehmen. Wir untersuchen das, indem wir die Zellen von gesunden Spendern in vitro in unterschiedlicher Weise stimulieren und mittels nachfolgender Proteomanalysen untersuchen, ob die Proteine gebildet wurden, die wir sonst nur in den Zellen aus erkrankten Patienten gefunden haben (Abb. 2). So konnten wir etwa feststellen, dass eine entzündliche Aktivierung zumindest einen Teil der ungewünschten Aktivitäten verursachen kann. Wenn wir einen Eindruck davon erhalten haben, welche Funktionen letztlich für den krankhaften Zustand verantwortlich sind, können wir gezielt auf die Suche nach Wirkstoffen gehen, die diese ungewünschten Funktionen unterdrücken. Hier zeigt sich jetzt ganz besonders die Nützlichkeit von umfassenden Proteomanalysen. Hat man erst einmal die relevanten Fehlfunktionen erkannt und analysiert, kann man Markerproteine bestimmen, welche diesen Zustand indizieren können. Diese Markerproteine können somit für diagnostische Zwecke herangezogen werden. Gleichzeitig können diese Markerproteine auch eingesetzt werden, um in In-vitro-Studien geeignete Wirkstoffe zu finden, welche diese ungewünschten Aktivitäten unterbinden können. Schließlich können dieselben Proteine auch analysiert werden, um im Patienten auch nachzuvollziehen, ob eine eingeleitete Therapie auch wirklich den gewünschten Erfolg erbringt. Nur wenn die Markerproteine im Blut des Patienten zurückgehen, hat das Medikament den gewünschten Erfolg erzielt.
Damit wird klar, dass eine genaue Bestimmbarkeit solcher Markerproteine im Blutserum eine Voraussetzung dafür ist, die oben skizzierte Strategie klinisch umzusetzen. Das ist keine triviale Aufgabe, weil die meisten dieser Markerproteine im Bereich von Pikogramm (10–12 g) pro Milliliter Serum zu erwarten sind. Wir leisten derzeit viel Aufwand, um aus einer Kombination von immunologischen Anreicherungsverfahren mit „targeted proteomics“ (selected reaction monitoring) diese methodische Hürde zu meistern. Sobald uns das aber gelingt, können wir individualisiert den funktionellen Zustand der an der Krankheit beteiligten Zellen aus Serumanalysen abschätzen, daraus einen optimierten Therapievorschlag erstellen und im weiteren Verlauf überprüfen, ob die Therapie die erwartete Wirkung aufweist.
Zusammenfassung und Ausblick
Das multiple Myelom könnte bei diesen Fragestellungen eine entscheidende Rolle spielen. Wenn wirklich, so wie von vielen Forschern heute vorgeschlagen, das Stroma eine entscheidende Rolle bei der Entwicklung von Tumoren hat5, so sollten wir auch das Stroma behandeln. Im MGUS-Patienten sind Tumorzellen bereits manifest, noch bevor die maligne Erkrankung ausgebrochen ist. Wir haben beobachtet, dass die Stromazellen auch hier schon entscheidend verändert sind. Vielleicht gelingt es uns, diese Veränderungen pharmakologisch zu korrigieren und damit den Ausbruch der Erkrankung des multiplen Myeloms zu verzögern oder vielleicht sogar zu verhindern. Wir haben in dieser Hinsicht noch viel Arbeit vor uns. Aber dann hätte Proteomik wohl tatsächlich einen bedeutenden Beitrag zur Entwicklung von neuen Therapien geleistet.
Danksagung: Mein Dank gilt allen Mitarbeitern dieses Projektes, insbesondere Prof. Johannes Drach und Dr. Astrid Slany. Einige der vorgestellten Konzepte wurden gemeinsam mit Dr. Verena Paulitschke (Abteilung für Dermatologie) im Rahmen eines gemeinsamen FWF-Projektes entwickelt. Die Abbildungen wurden mittels der von Dr. Johannes Griss programmierten Datenbank (GPDE) erstellt.
1 Anderson NL et al., A human proteome detection and quantitation project. Mol Cell Proteomics 2009; 8(5):883–6
2 Landgren O et al., Monoclonal gammopathy of undetermined significance (MGUS) consistently precedes multiple myeloma: a prospective study. Blood 2009; 113(22):5412–7
3 Chng WJ et al., Genetic events in the pathogenesis of multiple myeloma. Best Pract Res Clin Haematol 2007; 20(4):571–96
4 Griss J, Haudek-Prinz V, Gerner C, GPDE: A biological proteomic database for biomarker discovery and evaluation. Proteomics 2011; 11(5):1000–4
5 Bissell MJ, Hines WC, Why don’t we get more cancer? A proposed role of the microenvironment in restraining cancer progression. Nat Med 2011; 17(3):320–9